Слова «шипит» морфологический и фонетический разбор
Объяснение правил деление (разбивки) слова «шипит» на слоги для переноса.
Онлайн словарь Soosle.ru поможет: фонетический и морфологический разобрать слово «шипит» по составу, правильно делить на слоги по провилам русского языка, выделить части слова, поставить ударение, укажет значение, синонимы, антонимы и сочетаемость к слову «шипит».
Содержимое:
- 1 Слоги в слове «шипит»
- 2 Как перенести слово «шипит»
- 3 Морфемный разбор слова «шипит» по составу
- 4 Сходные по морфемному строению слова «шипит»
- 5 Синонимы слова «шипит»
- 6 Ударение в слове «шипит»
- 7 Фонетическая транскрипция слова «шипит»
- 8 Фонетический разбор слова «шипит» на буквы и звуки (Звуко-буквенный)
- 9 Предложения со словом «шипит»
- 10 Сочетаемость слова «шипит»
- 11 Значение слова «шипит»
- 12 Как правильно пишется слово «шипит»
Слоги в слове «шипит»
Количество слогов: 2
Как перенести слово «шипит»
ши—пит
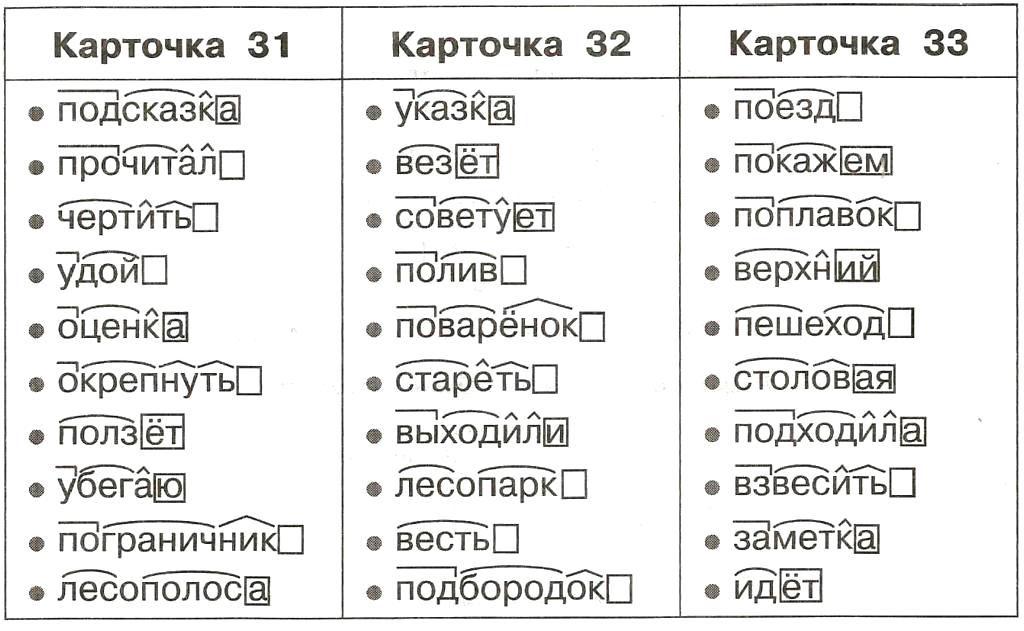
Морфемный разбор слова «шипит» по составу
| шип | корень |
| е | суффикс |
| ть | глагольное окончание |
шипеть
Сходные по морфемному строению слова «шипит»
Сходные по морфемному строению слова
Синонимы слова «шипит»
1. ворчать
ворчать
2. брюзжать
3. шикать
4. шептать
5. браниться
6. бурчать
Ударение в слове «шипит»
шипи́т — ударение падает на 2-й слог
Фонетическая транскрипция слова «шипит»
[шып’`ит]
Фонетический разбор слова «шипит» на буквы и звуки (Звуко-буквенный)
| Буква | Звук | Характеристики звука | Цвет |
|---|---|---|---|
| ш | [ш] | согласный, глухой парный, твёрдый, шипящий, шумный | ш |
| и | [ы] | гласный, безударный | и |
| п | [п’] | согласный, глухой парный, мягкий, шумный | п |
| и | [`и] | гласный, ударный | и |
| т | [т] | согласный, глухой парный, твёрдый, шумный | т |
Число букв и звуков:
На основе сделанного разбора делаем вывод, что в слове 5 букв и 5 звуков.
Буквы: 2 гласных буквы, 3 согласных букв.
Звуки: 2 гласных звука, 3 согласных звука.
Предложения со словом «шипит»
Попавший в петлю хвостоног, продолжал шипеть и шумно бороться с проволочной петлёй.Источник: Александр Редькин, Солнечный песок.
При попытке согнать её с гнезда гусыня начинаетИсточник: Н. М. Звонарев, Гуси, утки, индоутки. Прибыльная домашняя птицеферма от А до Я, 2010.
Парни уже перестали шипеть при каждом ударе, как коты, и только посапывали: видно, выдохлись.Источник: А. Н. Громов, Текодонт, 1990.
Сочетаемость слова «шипит»
1. змея шипит
2. вода шипела
3. кошка шипела
4. шипеть от боли
5. шипеть сквозь зубы
6. шипеть на кого-либо
7. (полная таблица сочетаемости)
Значение слова «шипит»
ШИПЕ́ТЬ , -плю́, -пи́шь; прич. наст. шипя́щий; несов. 1. Издавать глухие звуки, напоминающие протяжное произношение звука „ш“. (Малый академический словарь, МАС)
1. Издавать глухие звуки, напоминающие протяжное произношение звука „ш“. (Малый академический словарь, МАС)
Как правильно пишется слово «шипит»
Правописание слова «шипит»Орфография слова «шипит»
Правильно слово пишется:
Нумерация букв в слове
Номера букв в слове «шипит» в прямом и обратном порядке:
Опубликовано: 2020-05-18
Популярные слова
воспитанник , беседами , взбежавшие , взъерошив , выскребу , высчитанною , вытравлявшей , вячеславом , гемолизом , геннадиевичи , гимнастерочку , домоустройство , завибрируют , завинчивающимся , павлиньего , парабеллумами , парковавшемся , перебираемыми , плакатная , подающее , подлетать , подросту , положительнейшего , помпонах , поохотившимся , пражского , прогульном , прокашливаться , проституируя , противогазовые , развернувшее , разделе , раскрутилось , раскусывают , расторгну , резервированного , реорганизовавшем , респонсорною , сильванер , солея
Общесистемная разборка и сборка комплексов убиквитинлигазы SCF
1. Мурата С., Яширода Х., Танака К. Молекулярные механизмы сборки протеасом. Нац. Преподобный Мол. Клеточная биол. 2009; 10:104–115. doi: 10.1038/nrm2630. [PubMed] [CrossRef] [Google Scholar]
Мурата С., Яширода Х., Танака К. Молекулярные механизмы сборки протеасом. Нац. Преподобный Мол. Клеточная биол. 2009; 10:104–115. doi: 10.1038/nrm2630. [PubMed] [CrossRef] [Google Scholar]
2. Томко Р.Дж., Тейлор Д.В., Чен З.А., Ван Х.В., Раппсильбер Дж., Хохштрассер М. Одиночная альфа-спираль вызывает обширное ремоделирование крышки протеасомы и завершение сборки регуляторных частиц. . Клетка. 2015; 163:432–444. doi: 10.1016/j.cell.2015.090,022. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
3. Davis J.H., Tan Y.Z., Carragher B., Potter C.S., Lyumkis D., Williamson J.R. Модульная сборка бактериальной большой рибосомной субъединицы. Клетка. 2016;167:1610–1622.e15. doi: 10.1016/j.cell.2016.11.020. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
4. Сингх С., Ванден Брок А., Миллер Л., Чакер-Марго М., Клинге С. Созревание ядрышков процесса малых субъединиц человека. науч. Н. Ю. Н.Й. 2021; 373 doi: 10.1126/science.abj5338. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
5. Schnell H.M., Walsh R.M., Rawson S., Hanna J. Шаперон-опосредованная сборка ядра протеасомы — последние разработки и понимание структуры. Дж. Клеточная наука. 2022; 135 doi: 10.1242/jcs.259622. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Schnell H.M., Walsh R.M., Rawson S., Hanna J. Шаперон-опосредованная сборка ядра протеасомы — последние разработки и понимание структуры. Дж. Клеточная наука. 2022; 135 doi: 10.1242/jcs.259622. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
6. Pierce N.W., Lee J.E., Liu X., Sweredoski M.J., Graham R.L., Larimore EA, Rome M., Zheng N., Clurman B.E., Hess С. и др. Cand1 способствует сборке новых комплексов SCF за счет динамического обмена белками F-box. Клетка. 2013;153:206–215. doi: 10.1016/j.cell.2013.02.024. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
7. Земля А., Томас Ю., Кедзиора С., Кнебель А., Вуд Н.Т., Рабут Г., Курц Т. CSN- и CAND1-зависимое ремоделирование SCF-комплекса почкующихся дрожжей. Нац. коммун. 2013;4:1641. doi: 10.1038/ncomms2628. [PubMed] [CrossRef] [Google Scholar]
8. Wu S., Zhu W., Nhan T., Toth J.I., Petroski M.D., Wolf D.A. CAND1 контролирует in vivo динамику репертуара убиквитинлигазы cullin 1-RING. Нац. коммун. 2013;4:1642. doi: 10.1038/ncomms2636. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
коммун. 2013;4:1642. doi: 10.1038/ncomms2636. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
9. Reitsma J.M., Liu X., Reichermeier K.M., Moradian A., Sweredoski M.J., Hess S., Deshaies R.J. Состав и регуляция клеточного репертуара убиквитинлигаз SCF. Клетка. 2017;171:1326–1339.e14. doi: 10.1016/j.cell.2017.10.016. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
10. Liu X., Reitsma J.M., Mamrosh J.L., Zhang Y., Straube R., Deshaies R.J. Cand1-опосредованный механизм адаптивного обмена делает возможной вариацию экспрессии белка F-box. Мол. Клетка. 2018;69: 773–786.e6. doi: 10.1016/j.molcel.2018.01.038. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
11. Райхермайер К.М., Штраубе Р., Рейтсма Дж.М., Свередоски М.Дж., Роуз С.М., Морадян А., Ден Бестен В., Хинкль Т., Вершурен Э. ., Петцольд Г. и др. Анализ PIKES выявляет реакцию на деструкторы и ключевые регуляторные механизмы сети CRL4. Мол. Клетка. 2020;77:1092–1106.e9. doi: 10. 1016/j.molcel.2019.12.013. [PubMed] [CrossRef] [Google Scholar]
1016/j.molcel.2019.12.013. [PubMed] [CrossRef] [Google Scholar]
12. Энчев Р.И., Скотт Д.К., да Фонсека П.А., Шрайбер А., Монда Дж.К., Шульман Б.А., Питер М., Моррис Е.П. Структурная основа реципрокной регуляции между ФСК и ДНС. Cell Rep. 2012; 2: 616–627. doi: 10.1016/j.celrep.2012.08.019. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
13. Emberley E.D., Mosadeghi R., Deshaies R.J. Деконъюгация Nedd8 с Cul1 напрямую регулируется Skp1-F-box и субстратом, а сигналосома COP9 ингибирует денеддилированный SCF некаталитическим механизмом. Дж. Биол. хим. 2012; 287:29679–29689. doi: 10.1074/jbc.M112.352484. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
14. Фишер Э.С., Скрима А., Бём К., Мацумото С., Лингараю Г.М., Фати М., Ясуда Т., Кавадини С., Вакасуги М., Ханаока Ф. и др. Молекулярная основа архитектуры, нацеливания и активации убиквитинлигазы CRL4DDB2/CSA. Клетка. 2011; 147:1024–1039. doi: 10.1016/j.cell.2011.10.035. [PubMed] [CrossRef] [Google Scholar]
15. Cheng Y., Dai X., Zhao Y. AtCAND1, белок с повторами HEAT, который участвует в передаче сигналов ауксина у арабидопсиса. Завод Физиол. 2004; 135:1020–1026. doi: 10.1104/стр.104.044495. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Cheng Y., Dai X., Zhao Y. AtCAND1, белок с повторами HEAT, который участвует в передаче сигналов ауксина у арабидопсиса. Завод Физиол. 2004; 135:1020–1026. doi: 10.1104/стр.104.044495. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
16. Chuang H.W., Zhang W., Gray W.M. Arabidopsis ETA2, очевидный ортолог белка CAND1, взаимодействующего с куллином человека, необходим для ответов на ауксин, опосредованных убиквитинлигазой SCF(TIR1). Растительная клетка. 2004; 16:1883–189.7. doi: 10.1105/tpc.021923. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
17. Feng S.H., Shen Y.P., Sullivan J.A., Rubio V., Xiong Y., Sun T.P., Deng X.W. CAND1 арабидопсиса, немодифицированный CUL1-взаимодействующий белок, участвует во многих путях развития, контролируемых убиквитин/протеасом-опосредованной деградацией белка. Растительная клетка. 2004; 16: 1870–1882. doi: 10.1105/tpc.021949. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
18. Lo S.C., Hannink M. Опосредованная CAND1 рециркуляция адаптера субстрата необходима для эффективной репрессии Nrf2 с помощью Keap1. Мол. Клетка. биол. 2006; 26:1235–1244. doi: 10.1128/MCB.26.4.1235-1244.2006. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Опосредованная CAND1 рециркуляция адаптера субстрата необходима для эффективной репрессии Nrf2 с помощью Keap1. Мол. Клетка. биол. 2006; 26:1235–1244. doi: 10.1128/MCB.26.4.1235-1244.2006. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
19. Майор-Руис К., Ягер М.Г., Бауэр С., Бранд М., Син С., Ханзл А., Мюллер А.С., Менче Дж., Винтер Г.Е. Пластичность репертуара куллин-RING-лигазы формирует чувствительность к лиганд-индуцированной деградации белка. Мол. Клетка. 2019;75:849–858.e8. doi: 10.1016/j.molcel.2019.07.013. [PubMed] [CrossRef] [Google Scholar]
20. Виллемс А.Р., Шваб М., Тайерс М. Автостопщик по убиквитинлигазам куллина: SCF и его аналог. Биохим. Биофиз. Акта. 2004; 1695: 133–170. [PubMed] [Академия Google]
21. Сарикас А., Хартманн Т., Пан З.К. Семейство кулиновых белков. Геном биол. 2011;12:220. doi: 10.1186/gb-2011-12-4-220. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
22. Руснак Д.В., Чжэн Н. Структурная биология убиквитинлигаз CRL. Доп. Эксп. Мед. биол. 2020;1217:9–31. doi: 10.1007/978-981-15-1025-0_2. [PubMed] [CrossRef] [Google Scholar]
Доп. Эксп. Мед. биол. 2020;1217:9–31. doi: 10.1007/978-981-15-1025-0_2. [PubMed] [CrossRef] [Google Scholar]
23. Скаар Дж. Р., Паган Дж. К., Пагано М. Механизмы и функция рекрутирования субстрата белками F-бокса. Нац. Преподобный Мол. Клеточная биол. 2013;14:369–381. doi: 10.1038/nrm3582. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
24. Harper J.W., Schulman B.A. Регуляторные цепи убиквитинлигазы Cullin-RING: четверть века после гипотезы F-box. Анну. Преподобный Биохим. 2021; 90: 403–429. doi: 10.1146/annurev-biochem-0
-013613. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
25. Wang K., Deshaies RJ, Liu X. Сборка и регуляция убиквитинлигаз CRL. Доп. Эксп. Мед. биол. 2020;1217:33–46. дои: 10.1007/978-981-15-1025-0_3. [PubMed] [CrossRef] [Google Scholar]
26. Нгуен К.М., Бусино Л. Биология белков F-box: семейство SCF убиквитинлигаз E3. Доп. Эксп. Мед. биол. 2020;1217:111–122. doi: 10.1007/978-981-15-1025-0_8. [PubMed] [CrossRef] [Google Scholar]
27. Хао Б., Чжэн Н., Шульман Б.А., Ву Г., Миллер Дж.Дж., Пагано М., Павлетич Н.П. Структурные основы Cks1-зависимого распознавания p27(Kip1) убиквитинлигазой SCF(Skp2). Мол. Клетка. 2005; 20:9–19. [PubMed] [Академия Google]
Хао Б., Чжэн Н., Шульман Б.А., Ву Г., Миллер Дж.Дж., Пагано М., Павлетич Н.П. Структурные основы Cks1-зависимого распознавания p27(Kip1) убиквитинлигазой SCF(Skp2). Мол. Клетка. 2005; 20:9–19. [PubMed] [Академия Google]
28. Orlicky S., Tang X., Willems A., Tyers M., Sicheri F. Структурная основа выбора и ориентации фосфозависимого субстрата с помощью убиквитинлигазы SCFCdc4. Клетка. 2003; 112: 243–256. [PubMed] [Google Scholar]
29. Ву Г., Сюй Г., Шульман Б.А., Джеффри П.Д., Харпер Дж.В., Павлетич Н.П. Структура комплекса бета-TrCP1-Skp1-бета-катенин: связывание мотива разрушения и специфичность лизина убиквитинлигазы SCF (бета-TrCP1). Мол. Клетка. 2003; 11:1445–1456. [PubMed] [Академия Google]
30. Хао Б., Ольманн С., Сова М.Е., Харпер Дж.В., Павлетич Н.П. Структура комплекса Fbw7-Skp1-циклин E: распознавание мультисайтового фосфорилированного субстрата убиквитинлигазами SCF. Мол. Клетка. 2007; 26: 131–143. [PubMed] [Google Scholar]
31. Пан З.К., Кенцис А. , Диас Д.К., Ямоа К., Ву К. Недд8 о куллине: построение скоростной дороги к разрушению белка. Онкоген. 2004; 23:1985–1997. [PubMed] [Google Scholar]
, Диас Д.К., Ямоа К., Ву К. Недд8 о куллине: построение скоростной дороги к разрушению белка. Онкоген. 2004; 23:1985–1997. [PubMed] [Google Scholar]
32. Scott D.C., Свидерский В.О., Monda J.K., Lydeard J.R., Cho S.E., Harper J.W., Schulman B.A. Структура RING E3, пойманная в действие, раскрывает механизм лигирования убиквитин-подобного белка NEDD8. Клетка. 2014; 157:1671–1684. doi: 10.1016/j.cell.2014.04.037. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
33. Ляпина С., Коуп Г., Шевченко А., Серино Г., Цуге Т., Чжоу С., Вольф Д.А., Вэй Н., Шевченко А., Дешай Р.Дж. Продвижение расщепления конъюгата NEDD-CUL1 сигналосомой COP9. Наука. 2001; 292:1382–1385. [PubMed] [Google Scholar]
34. Борнштейн Г., Ганот Д., Хершко А. Регуляция неддилирования и денеддилирования cullin1 в убиквитинлигазе SCFSkp2 белком F-box и субстратом. проц. Натл. акад. науч. США. 2006;103:11515–11520. [Бесплатная статья PMC] [PubMed] [Google Scholar]
35. Lingaraju G.M., Bunker R. D., Cavadini S., Hess D., Hassiepen U., Renatus M., Fischer E.S., Thomä N.H. Кристаллическая структура сигналосомы COP9 человека. Природа. 2014; 512:161–165. doi: 10.1038/nature13566. [PubMed] [CrossRef] [Google Scholar]
D., Cavadini S., Hess D., Hassiepen U., Renatus M., Fischer E.S., Thomä N.H. Кристаллическая структура сигналосомы COP9 человека. Природа. 2014; 512:161–165. doi: 10.1038/nature13566. [PubMed] [CrossRef] [Google Scholar]
36. Мосадеги Р., Райхермайер К.М., Винклер М., Шрайбер А., Рейтсма Дж.М., Чжан Ю., Стенгель Ф., Цао Дж., Ким М., Свередоски М.Дж. , и другие. Структурный и кинетический анализ активации COP9-Signalosome и цикла денеддилирования убиквитинлигазы cullin-RING. электронная жизнь. 2016; 5 doi: 10.7554/eLife.12102. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
37. Cavadini S., Fischer E.S., Bunker R.D., Potenza A., Lingaraju G.M., Goldie K.N., Mohamed W.I., Faty M., Petzold G., Beckwith R.E., et al. Регуляция убиквитин-лигазы Cullin-RING сигналосомой COP9. Природа. 2016; 531: 598–603. doi: 10.1038/nature17416. [PubMed] [CrossRef] [Google Scholar]
38. Zheng J., Yang X., Harrell J.M., Ryzhikov S., Shim E.H., Lykke-Andersen K., Wei N., Sun H. , Kobayashi R., Zhang H. CAND1 связывается с ненедилированным CUL1 и регулирует образование комплекса убиквитин-Е3-лигазы SCF. Мол. Клетка. 2002;10:1519–1526. [PubMed] [Google Scholar]
, Kobayashi R., Zhang H. CAND1 связывается с ненедилированным CUL1 и регулирует образование комплекса убиквитин-Е3-лигазы SCF. Мол. Клетка. 2002;10:1519–1526. [PubMed] [Google Scholar]
39. Liu J., Furukawa M., Matsumoto T., Xiong Y. Модификация NEDD8 CUL1 диссоциирует p120(CAND1), ингибитор связывания CUL1-SKP1 и SCF-лигазы. Мол. Клетка. 2002; 10: 1511–1518. [PubMed] [Google Scholar]
40. Goldenberg S.J., Cascio T.C., Shumway S.D., Garbutt K.C., Liu J., Xiong Y., Zheng N. Структура комплекса Cand1-Cul1-Roc1 раскрывает регуляторные механизмы сборки мультисубъединичные куллин-зависимые убиквитинлигазы. Клетка. 2004;119: 517–528. [PubMed] [Google Scholar]
41. Schmidt M.W., McQuary P.R., Wee S., Hofmann K., Wolf D.A. Сборка и регулирование комплекса CRL под управлением F-box с помощью CSN и CAND1. Мол. Клетка. 2009; 35: 586–597. doi: 10.1016/j.molcel.2009.07.024. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
42. Zhang Y., Jost M., Pak R.A., Lu D. , Li J., Lomenick B., Garbis S.D., Li C.M., Weissman J.S. , Липфорд Дж.Р., Деше Р.Дж. Адаптивный обмен поддерживает сети убиквитинлигазы cullin-RING и правильное лицензирование репликации ДНК. проц. Натл. акад. науч. США. 2022;119doi: 10.1073/pnas.2205608119. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
, Li J., Lomenick B., Garbis S.D., Li C.M., Weissman J.S. , Липфорд Дж.Р., Деше Р.Дж. Адаптивный обмен поддерживает сети убиквитинлигазы cullin-RING и правильное лицензирование репликации ДНК. проц. Натл. акад. науч. США. 2022;119doi: 10.1073/pnas.2205608119. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
43. Schlierf A., Altmann E., Quancard J., Jefferson A.B., Assenberg R., Renatus M., Jones M., Hassiepen U., Шефер М., Кифф М. и др. Направленное ингибирование сигналосомы COP9 для лечения рака. Нац. коммун. 2016;7 doi: 10.1038/ncomms13166. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
44. Wiśniewski J.R., Hein M.Y., Cox J., Mann M. «Протеомная линейка» для определения числа копий белка и оценки концентрации без дополнительных стандартов. Мол. Клетка. Протеомика. 2014;13:3497–3506. doi: 10.1074/mcp.M113.037309. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
45. Чжэн Н., Шульман Б.А., Сонг Л., Миллер Дж.Дж., Джеффри П. Д., Ван П., Чу К., Кепп Д.М., Элледж С.Дж., Пагано М. и др. Структура убиквитинлигазного комплекса Cul1-Rbx1-Skp1-F boxSkp2 SCF. Природа. 2002; 416: 703–709. [PubMed] [Google Scholar]
Д., Ван П., Чу К., Кепп Д.М., Элледж С.Дж., Пагано М. и др. Структура убиквитинлигазного комплекса Cul1-Rbx1-Skp1-F boxSkp2 SCF. Природа. 2002; 416: 703–709. [PubMed] [Google Scholar]
46. Шульман Б.А., Каррано А.С., Джеффри П.Д., Боуэн З., Киннукан Э.Р., Финнин М.С., Элледж С.Дж., Харпер Дж.В., Пагано М., Павлетич Н.П. Взгляд на убиквитинлигазы SCF по структуре комплекса Skp1-Skp2. Природа. 2000;408:381–386. [PubMed] [Академия Google]
47. Мена Э.Л., Евтич П., Гребер Б.Дж., Джи К.Л., Лью Б.Г., Акопян Д., Ногалес Э., Куриян Дж., Рап М. Структурные основы контроля качества димеризации. Природа. 2020; 586: 452–456. doi: 10.1038/s41586-020-2636-7. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
48. Horn-Ghetko D., Krist D.T., Prabu J.R., Baek K., Mulder M.P.C., Klügel M., Scott D.C., Ovaa H., Kleiger Г., Шульман Б.А. Лигирование убиквитина с белками-мишенями F-box с помощью суперсборки SCF-RBR E3-E3. Природа. 2021;590: 671–676. doi: 10.1038/s41586-021-03197-9. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
[PMC free article] [PubMed] [CrossRef] [Google Scholar]
49. Ganoth D., Bornstein G., Ko T.K., Larsen B., Tyers M., Pagano M., Hershko A. Регулятор клеточного цикла белок Cks1 необходим для SCF(Skp2)-опосредованного убиквитинилирования p27. Нац. Клеточная биол. 2001; 3: 321–324. doi: 10.1038/35060126. [PubMed] [CrossRef] [Google Scholar]
50. Baek K., Krist D.T., Prabu J.R., Hill S., Klügel M., Neumaier L.M., von Gronau S., Kleiger G., Schulman B.A. NEDD8 образует поливалентную сборку лигирования убиквитина cullin-RING-UBE2D. Природа. 2020; 578: 461–466. doi: 10.1038/s41586-020-2000-y. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
51. Уинстон Дж.Т., Кепп Д.М., Чжу Ч.Х., Элледж С.Дж., Харпер Дж.В. Семейство белков F-box млекопитающих. Курс. биол. 1999;9:1180–11С3. doi: 10.1016/S0960-9822(00)80021-4. [PubMed] [CrossRef] [Google Scholar]
52. Welcker M., Wang B.Y., Rusnac D.V., Hussaini Y., Swanger J., Zheng N., Clurman B.E. Два дифосфорилированных дегрона контролируют деградацию c-Myc супрессором опухоли Fbw7. науч. Доп. 2022;8 doi: 10.1126/sciadv.abl7872. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
науч. Доп. 2022;8 doi: 10.1126/sciadv.abl7872. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
53. Йошида Ю., Тиба Т., Токунага Ф., Кавасаки Х., Иваи К., Сузуки Т., Ито Ю., Мацуока К., Йошида М., Танака К., Тай Т. Убиквитинлигаза Е3 который распознает сахарные цепи. Природа. 2002; 418:438–442. doi: 10.1038/nature00890. [PubMed] [CrossRef] [Google Scholar]
54. Мидзусима Т., Хирао Т., Ёсида Ю., Ли С.Дж., Чиба Т., Иваи К., Ямагути Ю., Като К., Цукихара Т., Танака K. Структурные основы убиквитинлигазы, узнающей сахар. Нац. Структура Мол. биол. 2004; 11: 365–370. [PubMed] [Академия Google]
55. Angers S., Li T., Yi X., MacCoss MJ, Moon R.T., Zheng N. Молекулярная архитектура и сборка убиквитинлигазы DDB1-CUL4A. Природа. 2006; 443: 590–593. [PubMed] [Google Scholar]
56. Cardote T.A.F., Gadd M.S., Ciulli A. Кристаллическая структура комплекса убиквитинлигазы Cul2-Rbx1-EloBC-VHL. Состав. 2017;25:901–911.e3. doi: 10.1016/j.str.2017.04.009. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
57. Дуда Д.М., Борг Л.А., Скотт Д.К., Хант Х.В., Хаммель М., Шульман Б.А. Структурное понимание активации NEDD8 лигаз кулин-RING: конформационный контроль конъюгации. Клетка. 2008;134:995–1006. [Бесплатная статья PMC] [PubMed] [Google Scholar]
Дуда Д.М., Борг Л.А., Скотт Д.К., Хант Х.В., Хаммель М., Шульман Б.А. Структурное понимание активации NEDD8 лигаз кулин-RING: конформационный контроль конъюгации. Клетка. 2008;134:995–1006. [Бесплатная статья PMC] [PubMed] [Google Scholar]
58. Yamoah K., Oashi T., Sarikas A., Gazdoiu S., Osman R., Pan Z.Q. Аутоингибиторная регуляция SCF-опосредованного убиквитинирования С-концевым хвостом человеческого куллина 1. проц. Натл. акад. науч. США. 2008;105:12230–12235. [Бесплатная статья PMC] [PubMed] [Google Scholar]
59. Saha A., Deshaies R.J. Мультимодальная активация убиквитинлигазы SCF путем конъюгации Nedd8. Мол. Клетка. 2008; 32:21–31. [Бесплатная статья PMC] [PubMed] [Google Scholar]
60. Fairweather S.J., Shah N., Brӧer S. Гетеромерные носители растворенных веществ: функция, структура, патология и фармакология. Доп. Эксп. Мед. биол. 2021; 21:13–127. doi: 10.1007/5584_2020_584. [PubMed] [CrossRef] [Google Scholar]
61. Эванс Р.М., Мангельсдорф Д.Дж. Ядерные рецепторы, RXR и Большой взрыв. Клетка. 2014; 157: 255–266. doi: 10.1016/j.cell.2014.03.012. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Клетка. 2014; 157: 255–266. doi: 10.1016/j.cell.2014.03.012. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
62. Ji AX, Privé G.G. Кристаллическая структура KLHL3 в комплексе с Cullin3. ПЛОС Один. 2013; 8 doi: 10.1371/journal.pone.0060445. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
63. Ким Ю.К., Квак М.Дж., Ку Б., Су Х.Ю., Джу К., Ли Дж., Юнг Дж.У., О Б.Х. Структурные основы межсубъединичного узнавания в комплексах элонгин BC-куллин 5-SOCS бокс-убиквитин-протеинлигаза. Акта Кристаллогр. Д биол. Кристаллогр. 2013;69:1587–1597. doi: 10.1107/S0907444913011220. [PubMed] [CrossRef] [Google Scholar]
64. Дуда Д.М., Ольшевский Дж.Л., Шуерманн Дж.П., Куринов И., Миллер Д.Дж., Нурс А., Альпи А.Ф., Шульман Б.А. Структура HHARI, убиквитинлигазы RING-IBR-RING: аутоингибирование E3 семейства Ариадны и понимание механизма лигирования. Состав. 2013;21:1030–1041. doi: 10.1016/j.str.2013.04.019. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
65. Scott D.C., Rhee D.Y., Duda D.M., Kelsall I.R., Olszewski J.L., Paulo J.A., de Jong A., Ovaa H., Alpi AF, Harper Дж.В., Шульман Б.А. Два различных типа лигаз E3 работают в унисон, чтобы регулировать убиквитилирование субстрата. Клетка. 2016;166:1198–1214.e24. doi: 10.1016/j.cell.2016.07.027. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Scott D.C., Rhee D.Y., Duda D.M., Kelsall I.R., Olszewski J.L., Paulo J.A., de Jong A., Ovaa H., Alpi AF, Harper Дж.В., Шульман Б.А. Два различных типа лигаз E3 работают в унисон, чтобы регулировать убиквитилирование субстрата. Клетка. 2016;166:1198–1214.e24. doi: 10.1016/j.cell.2016.07.027. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
66. Weissmann F., Petzold G., VanderLinden R., Huis In ‘t Veld P.J., Brown N.G., Lampert F., Westermann S., Stark H., Schulman B.A., Peters J.M. biGBac обеспечивает быструю сборку генов для экспрессии больших мультисубъединичных белковых комплексов. проц. Натл. акад. науч. США. 2016; 113:E2564–E2569. doi: 10.1073/pnas.1604935113. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
67. Xu L., Blackburn E.H. Белок Rif1 человека связывает аберрантные теломеры и выравнивается вдоль микротрубочек средней зоны анафазы. J. Cell Biol. 2004; 167:819–830. doi: 10.1083/jcb.200408181. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
68. Schindelin J., Arganda-Carreras I., Frize E., Kaynig V., Longair M., Pietzsch T., Preibisch S., Rueden К., Заальфельд С., Шмид Б. и соавт. Фиджи: платформа с открытым исходным кодом для анализа биологических изображений. Нац. Методы. 2012;9: 676–682. doi: 10.1038/nmeth.2019. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
Schindelin J., Arganda-Carreras I., Frize E., Kaynig V., Longair M., Pietzsch T., Preibisch S., Rueden К., Заальфельд С., Шмид Б. и соавт. Фиджи: платформа с открытым исходным кодом для анализа биологических изображений. Нац. Методы. 2012;9: 676–682. doi: 10.1038/nmeth.2019. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
Новые инструменты для автоматизированного определения структуры крио-ЭМ высокого разрешения в РЭЛИОН-3. электронная жизнь. 2018; 7 doi: 10.7554/eLife.42166. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
70. Рохоу А., Григорьев Н. CTFFIND4: быстрая и точная оценка расфокусировки по электронным микрофотографиям. J. Структура. биол. 2015;192: 216–221. doi: 10.1016/j.jsb.2015.08.008. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
71. Чжан К. Gctf: определение и коррекция CTF в реальном времени. J. Структура. биол. 2016; 193:1–12. doi: 10.1016/j.jsb.2015.11.003. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
72. Zheng S.Q., Palovcak E., Armache J.P., Verba K.A., Cheng Y., Agard D.A. MotionCor2: анизотропная коррекция движения, вызванного лучом, для улучшения криоэлектронной микроскопии. Нац. Методы. 2017;14:331–332. doi: 10.1038/nmeth.4193. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Zheng S.Q., Palovcak E., Armache J.P., Verba K.A., Cheng Y., Agard D.A. MotionCor2: анизотропная коррекция движения, вызванного лучом, для улучшения криоэлектронной микроскопии. Нац. Методы. 2017;14:331–332. doi: 10.1038/nmeth.4193. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
обучающее решение для крио-ЭМ объемной постобработки. коммун. биол. 2021;4:874. doi: 10.1038/s42003-021-02399-1. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
74. Бияни Н., Ригетто Р.Д., Маклеод Р., Кожоль-Берт Д., Кастано-Диез Д., Голди К.Н., Штальберг Х. Фокус: интерфейс между сбором данных и обработкой данных в крио-ЭМ. J. Структура. биол. 2017;198:124–133. doi: 10.1016/j.jsb.2017.03.007. [PubMed] [CrossRef] [Google Scholar]
75. Pettersen E.F., Goddard T.D., Huang C.C., Couch G.S., Greenblatt D.M., Meng E.C., Ferrin T.E. UCSF Chimera — система визуализации для поисковых исследований и анализа. Дж. Вычисл. хим. 2004; 25:1605–1612. doi: 10.1002/jcc.20084. [PubMed] [CrossRef] [Google Scholar]
[PubMed] [CrossRef] [Google Scholar]
76. Goddard T.D., Huang C.C., Meng E.C., Pettersen E.F., Couch G.S., Morris J.H., Ferrin T.E. UCSF ChimeraX: решение современных задач визуализации и анализа. Белковая наука. 2018;27:14–25. doi: 10.1002/pro.3235. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
77. Emsley P., Lohkamp B., Scott W.G., Cowtan K. Особенности и развитие лысухи. Акта Кристаллогр. Д биол. Кристаллогр. 2010;66:486–501. doi: 10.1107/S0907444910007493. [PMC free article] [PubMed] [CrossRef] [Google Scholar]
78. Афонин П.В., Клахольц Б.П., Мориарти Н.В., Пун Б.К., Соболев О.В., Тервиллигер Т.С., Адамс П.Д., Уржумцев А. Новые инструменты для анализа и валидации крио-ЭМ карт и атомных моделей. Акта Кристаллогр. D Структура. биол. 2018;74:814–840. дои: 10.1107/S2059798318009324. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
79. Scott D.C., Schulman B.A. Двухцветные анализы убиквитинирования с отслеживанием импульсов для одновременного мониторинга затравки и удлинения субстрата. Методы Энзимол. 2019; 618: 29–48. doi: 10.1016/bs.mie.2019.01.004. [PubMed] [CrossRef] [Google Scholar]
Методы Энзимол. 2019; 618: 29–48. doi: 10.1016/bs.mie.2019.01.004. [PubMed] [CrossRef] [Google Scholar]
80. Кулак Н.А., Пихлер Г., Парон И., Нагарадж Н., Манн М. Минимальная обработка инкапсулированного протеомного образца для оценки числа копий в эукариотических клетках. Нац. Методы. 2014;11:319–324. doi: 10.1038/Nmeth.2834. [PubMed] [CrossRef] [Google Scholar]
81. Брудерер Р., Бернхардт О.М., Ганди Т., Миладинович С.М., Ченг Л.Ю., Месснер С., Эренбергер Т., Занотелли В., Бутшайд Ю., Эшер С. , и другие. Расширение границ количественного профилирования протеома за счет независимого от данных сбора данных и применения к трехмерным микротканям печени, обработанным ацетаминофеном. Мол. Клетка. Протеомика. 2015;14:1400–1410. doi: 10.1074/mcp.M114.044305. [Бесплатная статья PMC] [PubMed] [CrossRef] [Google Scholar]
Напечатайте свои собственные изображения, используя куркуму и солнечный свет.
Куркума — одна из самых ярких специй, которую вы когда-либо найдете, и основной продукт индийской кухни, но она может сделать гораздо больше, чем придать вашей еде аромат. Куркумин, одно из основных соединений в растении, имеет множество преимуществ для здоровья, и люди веками использовали его для окрашивания тканей.
Куркумин, одно из основных соединений в растении, имеет множество преимуществ для здоровья, и люди веками использовали его для окрашивания тканей.
Но куркумин также является фотосенсибилизатором, то есть он химически реагирует при воздействии света. На самом деле, вы можете использовать его для печати изображений, используя технику, называемую антотипией, которая была разработана в 19 веке.го века и использует растительные эмульсии и воздействие солнца для создания изображений на пористых поверхностях, таких как бумага и ткань.
Как создать антотип своими руками в домашних условиях
Статистика
Время: 10 минут (подготовка), от 1 до 3 часов (экспозиция)
Стоимость материала: менее 25 центов за отпечаток
Сложность: Easy
Материалы
- 1 чайная ложка порошка куркумы
- 2 чайные ложки буры
- ⅕ стакана или 50 миллилитров медицинского спирта
- ½ стакана воды
- 1 лист волокнистой бумаги
- Негатив, то есть то, что вы хотите напечатать — это может быть цветок, лист или что-то еще
Инструменты
- Два небольших контейнера
- Ложка
- Губка (или кисть)
- Распылитель
- Кофейный фильтр (или марля)
- Мерная ложка
- Мерные стаканы
- Фоторамка
- Солнце
- Бумажные полотенца
- (дополнительно) Резиновые перчатки
Инструкции
1. Смешайте порошок куркумы и спирт в контейнере. Куркума окрашивает почти все, к чему прикасается, поэтому мы рекомендуем защищать руки резиновыми перчатками и накрывать одежду фартуком или даже мешком для мусора. Куркума также окрашивает пластиковые контейнеры, поэтому лучше использовать одноразовые или стеклянные контейнеры.
Смешайте порошок куркумы и спирт в контейнере. Куркума окрашивает почти все, к чему прикасается, поэтому мы рекомендуем защищать руки резиновыми перчатками и накрывать одежду фартуком или даже мешком для мусора. Куркума также окрашивает пластиковые контейнеры, поэтому лучше использовать одноразовые или стеклянные контейнеры.
2. Процедить смесь через кофейный фильтр или марлю в другую емкость. Это позволит избавиться от порошка, что позволит красящему спирту более равномерно растечься по бумаге.
[См. также: Эта слизь, сделанная своими руками, может очистить самые грязные части вашего дома и автомобиля]
3. В самом темном месте, которое у вас есть, нанесите краску на бумагу и дайте ей высохнуть. Свет запускает процесс обесцвечивания красителя, который вам нужен только тогда, когда вы экспонируете свое изображение. Тем не менее, это не обязательно должно быть кромешной тьмой — шкаф, кладовая или полутемный кухонный уголок вполне подойдут. Нанесите ровный слой краски на бумагу, следя за тем, чтобы цвет был как можно более ровным. Когда закончите, подождите около 15 минут, пока бумага высохнет.
Нанесите ровный слой краски на бумагу, следя за тем, чтобы цвет был как можно более ровным. Когда закончите, подождите около 15 минут, пока бумага высохнет.- Примечание: Если вы делаете это в шкафу, обязательно защитите хранящуюся одежду, так как краска куркумы может разбрызгаться и испортить ее. Если вы используете плоскую поверхность, например поднос, для сушки бумаги, рекомендуется сначала положить слой бумажных полотенец, чтобы они впитали излишки красителя.
- Совет: Чем больше волокон в бумаге, тем лучше. Лучше всего подойдет переработанная или акварельная бумага, но вы можете использовать все, что у вас есть, включая бумагу для принтера формата A4.
4. Поместите объект отпечатка (негатив) на бумагу. Здесь вы можете быть настолько изобретательны, насколько захотите, так что выбирайте все, что хотите, чтобы оставить отпечаток на бумаге. Это может быть что-то простое, вроде прижатых цветов или листьев, или сложное, например, изображение, напечатанное на прозрачной пленке или нанесенное архивными чернилами на тонкую бумагу. Если вы используете печатный или трассированный негатив, на нем должны быть толстые, жирные линии, чтобы заблокировать как можно больше света.
Если вы используете печатный или трассированный негатив, на нем должны быть толстые, жирные линии, чтобы заблокировать как можно больше света.
- Совет для профессионалов: Вы можете попробовать использовать настоящую фотографию в качестве негатива — просто распечатайте нужную черно-белую фотографию на домашнем принтере. Имейте в виду, что обычная бумага для принтера блокирует часть света, поэтому вам, вероятно, придется подождать дольше, пока ваш антотип будет готов.
5. Когда композиция вас устроит, поместите ее в фоторамку. Если у вас нет рамки нужного размера, не беспокойтесь. Технически вам нужно только стекло, так как оно действует как груз, удерживая все на месте, пропуская свет. Например, если у вас есть большая рамка, которую вы можете разобрать, вы можете использовать ее, чтобы показать столько изображений, сколько поместится под ней.
6. Выставьте изображения на солнце и подождите. Разместите изображения на ровной поверхности под солнцем. Время, в течение которого вы их не используете, будет зависеть от времени суток, погоды и уровня контраста, который вы хотите придать своему изображению. Если выпал пасмурный день, подумайте о дополнительном времени экспозиции.
Время, в течение которого вы их не используете, будет зависеть от времени суток, погоды и уровня контраста, который вы хотите придать своему изображению. Если выпал пасмурный день, подумайте о дополнительном времени экспозиции.
[См. также: Как приготовить овсяное молоко — с помощью науки]
Насколько хорошо ваш негатив блокирует свет, также является важным фактором. Физические барьеры, такие как зеленые листья, оставят четкий темный отпечаток, в то время как изображение с плохо пигментированными линиями будет пропускать свет, в результате чего линии будут слабыми или даже отсутствующими. Вы можете поэкспериментировать с разным временем выдержки и углами по отношению к солнцу — просто убедитесь, что вы не двигаете свой негатив, так как будет сложно поместить его обратно в то же самое положение, и вы получите размытое изображение. изображение.
Это полностью экспонированный антотип по сравнению с окрашенным куркумином листом бумаги до экспонирования. Sandra Gutierrez G.
Sandra Gutierrez G. - Профессиональный совет: Оценка цвета бумаги — хороший способ определить, готово ли ваше изображение. Вы хотите, чтобы открытая поверхность превратилась из ярко-желтой в бледно-лимонный оттенок.
7. Смешайте раствор буры. Просто добавьте буру и воду в пульверизатор.
- Профессиональный совет: Используйте теплую воду, чтобы полностью растворить буру.
8. По истечении времени выдержки переместите изображения подальше от солнечного света. Вы заметите, что цвет фона вашего изображения стал намного светлее, чем раньше. Как только вы удалите негатив, вы также заметите отпечаток, который он оставил в гораздо более темном цвете.
- Примечание: Если вам нравится этот вид, вы можете остановиться здесь и выставить свой новый отпечаток на обозрение. Проблема в том, что процесс обесцвечивания не остановится, и ваш образ со временем потускнеет.

9. Закрепите изображение тонким слоем раствора буры. Положите отпечаток на бумажное полотенце. Хорошо встряхните раствор буры и распылите его тонким слоем на отпечаток. Линии темно-желтого цвета приобретут различные оттенки оранжевого, красного и коричневого в течение 10 минут.
- Примечание: Не теряйте терпения — распыление большего количества раствора буры также вызовет реакцию фона, оставив большое коричневое пятно.
10. Дайте изображению высохнуть. Как и любой творческий метод, антотипия не является совершенной наукой, и вам, вероятно, придется поэкспериментировать, прежде чем вы получите именно то, что хотите. Попробуйте разные концентрации красителя и разные негативы. Может быть, вы посмотрите, как лишний час экспозиции влияет на ваше изображение, или влияет ли место, которое вы используете для экспонирования своих фотографий, на ваши конечные результаты. Это часть развлечения.
Наука об антотипе
Солнечный свет — это волна, но это также и энергия в форме фотонов.